当社社員名(赤字)が記載された特許・論文・出版物です。
AI・機械学習 (創薬、データ解析)
創薬における機械学習と分子シミュレーション:生体での膜透過現象 真下 忠彰, 中村 寛則, 若林 良徳, 黒澤 隆, 福西 快文. アンサンブル : 分子シミュレーション研究会会誌 Vol. 21, No.2, April 2019 (通巻86号)
Prediction of Protein compound Binding Energies from Known Activity Data: Docking-score-based Method and its Applications., Yoshifumi Fukunishi, Yasunobu Yamashita, Tadaaki Mashimo, and Haruki Nakamura. Mol. Inf. doi:10.1002/minf.201700120 (2018 Feb.)
データベース利用での分子シミュレーション:半シミュレーション的学習法 (特集 創薬で活躍する分子シミュレーション) 福西快文, 若林良徳, 真下忠彰. アンサンブル : 分子シミュレーション研究会会誌 19(4), 245-255, 2017-10
An Application of Deep Learning for Classifying Chemical Structures. Yoshinori Wakabayashi, Tadaaki Mashimo, Mitsuhito Wada. CBI学会2016年大会 ポスター P2-28 (2016)
創薬 (溶解度予測など)
Structural Fluctuations of Aromatic Residues in an Apo-Form Reveal Cryptic Binding Sites: Implications for Fragment-Based Drug Design, Iida S, Nakamura HK, Mashimo T, Fukunishi Y, J Phys Chem B. 2020 Nov 3. (2020)
A2B adenosine receptor inhibition by the dihydropyridine calcium channel blocker nifedipine involves colonic fluid secretion, Asano T, Noda Y, Tanaka KI, Yamakawa N, Wada M, Mashimo T, Fukunishi Y, Mizushima T, Takenaga M, Sci Rep. 10: 3555 (2020)
Prediction of Passive Membrane Permeability by Semi-Empirical Method Considering Viscous and Inertial Resistances and Different Rates of Conformational Change and Diffusion, Fukunishi Y, Mashimo T, Yoshifumi Fukunishi, Kurosawa T, Wakabayashi Y, Nakamura HK, Takeuchi K, Mol Inform. 15: 0 (2019)
Quantitative analysis of aggregation-solubility relationship by in-silico solubility prediction, Tadaaki Mashimo, Yoshifumi Fukunishi, Masaya Orita, Naoko Katayama, Shigeo Fujita, Haruki Nakamura, International Journal of High Throughput Screening, 2010:1, 99-107
Quantitative analysis of aggregation-solubility relationship by in-silico solubility prediction(溶解度予測による凝集-溶解度関係の定量解析), Tadaaki Mashimo, Yoshifumi Fukunishi, Masaya Orita, Naoko Katayama, Shigeo Fujita, Haruki Nakamura, CBRC2010ポスター (ポスター賞)
Quantitative analysis of aggregation-solubility relationship by in-silico solubility prediction, Tadaaki Mashimo, Yoshifumi Fukunishi, Masaya Orita, Naoko Katayama, Shigeo Fujita and Haruki Nakamura, CBI Annual Meeting 2010 ポスターP7-04(優秀賞)
Aqueous solubility prediction by a neural network model with using calculated physical properties from 3D structures of molecule(分子の3D構造から算出された物理的性質を用いたニューラルネットワークモデルによる水溶解度推定), Mashimo Tadaaki, Fukunishi Yoshifumi, Orita Masaya, 情報計算化学生物学会大会予稿集 2008 pp.80
クラウド (創薬、データ解析)
MEGADOCK-Azure: High-performance protein-protein interaction prediction system on Microsoft Azure HPC. Masahito Ohue, Yuki Yamamoto, Hiroyuki Sato, Takashi Matsushita and Yutaka Akiyama. 第5回生命医薬情報学連合大会(IIBMP 2016)ポスター 52 ポスター賞受賞 (2016)
Scalability and performance of applications on cloud computing: virtual screenings and MD simulations of myPresto suite. Tadaaki Mashimo, Mitsuhito Wada, Yoshinori Wakabayashi, Yoshifumi Fukunishi and Haruki Nakamura. 第5回生命医薬情報学連合大会(IIBMP 2016)ポスター 60 (2016)
バイオデータベースの統合化
Mikan genome database (MiGD): integrated database of genome annotation, genomic diversity, and CAPS marker information for mandarin molecular breeding, Yoshihiro Kawahara, Tomoko Endo, Mitsuo Omura, Yumiko Teramoto, Takeshi Itoh, Hiroshi Fujii, Takehiko Shimada,
jsbbs.19097, 2020.
SOGO: A Novel Research Framework for Crop and Livestock Genome Studies, Koji Doi, Elena Solovieva, Yumiko Teramoto, Tomomi Ishikawa, Hiroshi Yasue, Akitoshi Goto, Akio Miyao,
Plant and Animal Genome Conference (PAG XXIV), P1148, 2015.1
SOGO: 農畜産物ゲノム研究のためのデータベース統合と解析環境の整備
土井考爾、ソロビヨワ・イェレナ、寺本由美子、石川友美、後藤明俊、宮尾安藝雄、トーゴーの日シンポジウム2014
RDFによるデータ統合と相互運用性のための技術開発
片山俊明、西澤達也、三嶋博之、川島秀一、岡本忍、藤澤貴智、トーゴーの日シンポジウム2014
TogoWS RESTサービスによるUCSCゲノムデータベースの利用, 三嶋博之, 西澤達也, 吉浦孝一郎, 片山俊明, 日本人類遺伝学会大会プログラム・抄録集 巻:58th ページ:130, 2013
The 2nd DBCLS BioHackathon: interoperable bioinformatics Web services for integrated applications, Toshiaki Katayama, Mark D Wilkinson, Rutger Vos, Takeshi Kawashima, Shuichi Kawashima, Mitsuteru Nakao, Yasunori Yamamoto, Hong-Woo Chun, Atsuko Yamaguchi, Shin Kawano, Jan Aerts, Kiyoko F Aoki-Kinoshita, Kazuharu Arakawa, Bruno Aranda, Raoul JP Bonnal, Jose M Fernandez, Takatomo Fujisawa, Paul MK Gordon, Naohisa Goto, Syed Haider, Todd Harris, Takashi Hatakeyama, Isaac Ho, Masumi Itoh, Arek Kasprzyk, Nobuhiro Kido, Young-Joo Kim, Akira R Kinjo, Fumikazu Konishi, Yulia Kovarskaya, Greg von Kuster, Alberto Labarga, Vachiranee Limviphuvadh, Luke McCarthy, Yasukazu Nakamura, Yunsun Nam, Kozo Nishida, Kunihiro Nishimura, Tatsuya Nishizawa, Soichi Ogishima, Tom Oinn, Shinobu Okamoto, Shujiro Okuda, Keiichiro Ono, Kazuki Oshita, Keun-Joon Park, Nicholas Putnam, Martin Senger, Jessica Severin, Yasumasa Shigemoto, Hideaki Sugawara, James Taylor, Oswaldo Trelles, Chisato Yamasaki, Riu Yamashita, Noriyuki Satoh and Toshihisa Takagi, Journal of Biomedical Semantics 2011, 2:4 doi:10.1186/2041-1480-2-4
TogoDB embedded: sneak preview – 次世代TogoDBが遍在する仕組み, 片山俊明, 中尾光輝, 西澤達也, 第14回オープンバイオ研究会, 2011-3-26
The DBCLS BioHackathon: standardization and interoperability for bioinformatics web services and workflows. Toshiaki Katayama, Kazuharu Arakawa, Mitsuteru Nakao, Keiichiro Ono, Kiyoko F Aoki-Kinoshita, Yasunori Yamamoto, Atsuko Yamaguchi, Shuichi Kawashima, Hong-Woo Chun, Jan Aerts, Bruno Aranda, Lord H Barboza, Raoul JP Bonnal, Richard Bruskiewich, Jan C Bryne, Jose M Fernandez, Akira Funahashi, Paul MK Gordon, Naohisa Goto, Andreas Groscurth, Alex Gutteridge, Richard Holland, Yoshinobu Kano, Edward A Kawas, Arnaud Kerhornou, Eri Kibukawa, Akira R Kinjo, Michael Kuhn, Hilmar Lapp, Heikki Lehvaslaiho, Hiroyuki Nakamura, Yasukazu Nakamura, Tatsuya Nishizawa, Chikashi Nobata, Tamotsu Noguchi, Thomas M Oinn, Shinobu Okamoto, Stuart Owen, Evangelos Pafilis, Matthew Pocock, Pjotr Prins, Rene Ranzinger, Florian Reisinger, Lukasz Salwinski, Mark Schreiber, Martin Senger, Yasumasa Shigemoto, Daron M Standley, Hideaki Sugawara, Toshiyuki Tashiro, Oswaldo Trelles, Rutger A Vos, Mark D Wilkinson, William York, Christian M Zmasek, Kiyoshi Asai and Toshihisa Takagi, Journal of Biomedical Semantics 2010, 1:8 doi:10.1186/2041-1480-1-8
TogoWS: integrated SOAP and REST APIs for interoperable bioinformatics Web services, Toshiaki Katayama, Mitsuteru Nakao and Toshihisa Takagi, (Acknowledgements: Tatsuya Nishizawa), Nucl. Acids Res. (2010) 38 (suppl 2): W706-W711.
創薬 (ドッキング/化合物スクリーニング)
Development of protein-ligand docking engine sievgene_M for manycore processors. (メニーコアプロセッサ向け蛋白質・リガンドドッキングエンジン sievgene_M の開発) Takanori Sugihara, Takashi Kurosawa, Hironori Nakamura, Tadaaki Mashimo, Yoshifumi Fukunishi, Haruki Nakamura. 生物物理(Web) 巻:57 号:Supplement 1-2 ページ:S206 (WEB ONLY) (2017)
Human insight in drug discovery: docking pose selection in protein-ligand docking and hit-compounds selection in structure-based virtual screening. Kazuki Yamamoto, Yoshitaka Moriwaki, Keita Oda, Masahito Ohue, Itsuo Nakane, Ryunosuke Yoshino, Hayase Hakariya, Tadaaki Mashimo, Mitsuhito Wada, Yoshifumi Fukunishi. CBI学会2016年大会 ポスター P2-14 Excellent Poster (2016)
Towards Desktop laboratory from MD, docking, and virtual screening to bio assay: Computation performance. Tadaaki Mashimo, Mitsuhito Wada, Yoshinori Wakabayashi, Yoshifumi Fukunishi, Haruki Nakamura. CBI学会2016年大会 ポスター P2-18 (2016)
Human insight in drug discovery: docking pose selection in protein-ligand docking and hit-compounds selection in structure-based virtual screening. Kazuki Yamamoto, Yoshitaka Moriwaki, Keita Oda, Masahito Ohue, Itsuo Nakane, Ryunosuke Yoshino, Hayase Hakariya, Tadaaki Mashimo, Mitsuhito Wada and Yoshifumi Fukunishi. 第5回生命医薬情報学連合大会(IIBMP 2016)ポスター 64 (2016)
Miscellaneous Topics in Computer-Aided Drug Design: Synthetic Accessibility and GPU Computing, and Other Topics. Yoshifumi Fukunishi, Tadaaki Mashimo, Kiyotaka Misoo, Yoshinori Wakabayashi, Toshiaki Miyaki, Seiji Ohta, Mayu Nakamura, Kazuyoshi Ikeda, Curr Pharm Des. 2016 ;22(23):3555-68
創薬支援総合ソフトウェアmyPresto:製品展開と産業応用 (Drug development software myPresto: it’s commercial products and applications). 福西 快文, 真下 忠彰, 和田 光人, 第15回 産総研・産技連 LS-BT合同研究発表会「ビッグデータとビッグシミュレーションによる生命医科学の未来」(理研・産総研共同シンポジウム)ポスターP043 (2016)
Integrated In-Silico Drug Screening System for G-Protein-Coupled Receptors by Using Universal Active Probes (普遍的活性プローブを用いたG蛋白質共役受容体に対する統合in‐silicoスクリーニングシステム), WADA Mitsuhito, KANAMORI Eiji, MASHIMO Tadaaki, FUKUNISHI Yoshifumi, NAKAMURA Haruki, 情報計算化学生物学会大会予稿集 2011, CBI-P1-17
In silico fragment screening by replica generation (FSRG) method for fragment-based drug design, Yoshifumi Fukunishi, Tadaaki Mashimo, Masaya Orita, Kazuki Ohno, and Haruki Nakamura, Journal of chemical information and modeling, Vol. 49, No. 4. (2009), pp.925-933
OdaibaDock: Protein-Protein docking system using a high-performance FFT algorithm on BlueGene/L(48. Bioinformatics, genomics and proteomics (II),Poster Session,Abstract,Meeting Program of EABS & BSJ 2006), Yoshikawa Tatsuya, Tsukamoto Koki, Hourai Yuichiro, Mashimo Tadaaki, Akiyama Yutaka, 生物物理 46(SUPPLEMENT_2), S407, 2006
超並列計算機BlueGene(BlueProtein)を用いた大規模タンパク質ドッキングシステムの開発、 吉川 達也, 塚本 弘毅, 蓬来 祐一郎, 真下 忠彰, 秋山 泰.、 CBRC 2006 予稿集 G3
分子動力学・MPI/GPU高速計算
分子動力学シミュレーション解析人工知能システムの応用研究、 山根 努, 三十尾 潔高、 独立行政法人 理化学研究所 情報システム部 2022年度利用報告書(HOKUSAI)
Variation of free-energy landscape of the p53 C-terminal domain induced by acetylation: Enhanced conformational sampling, Shinji Iida, Tadaaki Mashimo, Takashi Kurosawa, Hironobu Hojo, Hiroya Muta, Yuji Goto, Yoshifumi Fukunishi, Haruki Nakamura, Junichi Higo, Journal of Computational Chemistry 2016, 37 (31), 2687-2700
myPresto/omegagene: a GPU-accelerated molecular dynamics simulator tailored for enhanced conformational sampling methods with a non-Ewald electrostatic scheme, Kota Kasahara, Benson Ma, Kota Goto, Bhaskar Dasgupta, Junichi Higo, Ikuo Fukuda, Tadaaki Mashimo, Yutaka Akiyama, Haruki Nakamura, Biophysics and Physicobiology, 13: 209-216, 2016.
Elastic properties of dynein motor domain obtained from all-atom molecular dynamics simulations., Kamiya N, Mashimo T, Takano Y, Kon T, Kurisu G, Nakamura H.,Protein Eng Des Sel. 2016 Aug;29(8):317-25.
Virtual-system-coupled adaptive umbrella sampling to compute free-energy landscape for flexible molecular docking, Junichi Higo, Bhaskar Dasgupta, Tadaaki Mashimo, Kota Kasahara, Yoshifumi Fukunishi, Haruki Nakamura, J. Comput. Chem. 36, 1489-1501 (2015).
Molecular Dynamics Simulation Accelerated by GPU for GPCR with non-Ewald Algorithm, Tadaaki Mashimo, Takayuki Kochi, Yoshifumi Fukunishi, Narutoshi Kamiya, Yu Takano, Ikuo Fukuda, Haruki Nakamura,TSUBAME ESJ. 10, 25-29 (2013)
Molecular dynamics simulations accelerated by GPU for biological macromolecules with a non-Ewald scheme for electrostatic interactions,Tadaaki Mashimo, Yoshifumi Fukunishi, Narutoshi Kamiya, Yu Takano, Ikuo Fukuda, Haruki Nakamura,Journal of Chemical Theory and Computation,2013, 9 (12), pp 5599-5609
All-atomic molecular dynamics simulations of dynein motor domain in explicit water, KAMIYA Narutoshi, MASHIMO Tadaaki, TAKANO Yu, KON Takahide, KURISU Genji, 日本蛋白質科学会年会予稿集 2013, 13, pp.95
GPU-accelerated calculation of electrostatic interactions with zero-dipole summation method, Tadaaki Mashimo, Yoshifumi Fukunishi, Ikuo Fukuda, Narutoshi Kamiya, Yu Takano, Haruki Nakamura, 情報処理学会 ハイパフォーマンスコンピューティングと計算科学シンポジウム論文集 2013 pp.70
A simple non-Ewald scheme for calculating electrostatic interactions of charged particle systems: the zero-dipole summation method and the application to molecular systems, Ikuo Fukuda, Narutoshi Kamiya, Takamasa Arakawa, Tadaaki Mashimo, Yu Takano, Haruki Nakamura, 情報処理学会 ハイパフォーマンスコンピューティングと計算科学シンポジウム論文集 2013 pp.73
GPUを利用したZero-dipole summation法による静電相互作用計算の高速化、 真下 忠彰, 福西 快文,福田 育夫,神谷 成敏,鷹野 優,中村 春木、 HPCS2013(情報処理学会 2013年ハイパフォーマンスコンピューティングと計算科学シンポジウム)ポスターP1-9
GPU を用いた生体高分子の高速分子動力学計算、 中村 春木, 肥後 順一, 神谷 成敏, 笠原 浩太, 真下 忠彰、 独立行政法人 理化学研究所 情報基盤センター 平成 25 年度 RICC 利用報告書
大規模並列化に適したMDプログラム開発、 大野 洋介,小山 洋,長谷川 亜樹,舛本 現,似鳥 啓吾,古石 貴裕,森本 元太郎,吉田 正典,田附 八束,熊木 竜也,来栖 瑛佑,ノヴィッチコフ グレブ,荒川 貴道,三十尾 潔高,吉川 広幸、 独立行政法人 理化学研究所 情報基盤センター 平成 23 年度 RICC 利用報告書
Binding Free Energy Analysis based on GPGPU cluster, M. Sekijima, K. Misoo, K. Ohno, M. Orita, 24th Annual Symposium of the The Protein Society, 2010, San Diego
Development of Molecular Dynamics Programs for Protein with a ParallelizedBarnes-Hut Code, Kiyotaka Misoo, Yutaka Akiyama, Yoshihisa Shizawa, and Minoru Saito, Proc. of the Fourth International Conference on High-Performance Computing in Asia-Pacific Region (HPC-Asia 2000), Vol.2, pp.1103-1111
並列化Barnes-Hut tree codeを用いた高精度なタンパク質分子動力学法プログラムの開発(<特集>並列処理), 三十尾 潔高, 志澤 由久, 秋山 泰, 斎藤 稔, 情報処理学会論文誌, 2000, Vol.41, No.5, pp.1538-1548
並列化Barnes-Hut tree codeを用いた高精度なタンパク質分子動力学法プログラムの開発, 三十尾潔高, 志澤由久, 秋山 泰, 斎藤 稔, 並列処理シンポジウムJSPP’99 論文集, pp.143-150, Jun.1999.
分子動力学法プログラムAMBERとBarnes-Hut tree codeの並列化による高速化(<特集>並列処理), 斎藤 稔 , 三十尾 潔高, 志澤 由久, 丸川 一志 , 秋山 泰 , 野口 保 , 鬼塚 健太郎, 情報処理学会論文誌, 1999, Vol. 40, No. 5, pp.2142-2151
分子動力学法プログラムAMBERとBarnes-Hut tree codeの並列化による高速化, 斉藤 稔, 三十尾潔高, 志澤由久, 丸川一志, 秋山 泰, 野口 保, 鬼塚健太郎, 並列処理シンポジウム JSPP’98論文集, pp.231-238, Oct.1998.
分子動力学法プログラムAMBERとBarnes-Hut tree codeの並列化と性能評価, 斎藤 稔, 三十尾 潔高, 志澤 由久, 丸川 一志, 秋山 泰, 野口 保, 鬼塚 健太郎, 情報処理学会研究報告. [ハイパフォーマンスコンピューティング] 97(121), pp.61-66, 12 Dec., 1997.
タンパク質解析
PSCDB: a database for protein structural change upon ligand binding. Amemiya T, Koike R, Kidera A, Ota M. (Acknowledgements: Kiyotaka Misoo), Nucleic Acids Research, 2012 Jan;40(1):D554-8. Epub 2011 Nov 10.
SAHG, a comprehensive database of predicted structures of all human proteins. Motono C, Nakata J, Koike R, Shimizu K, Shirota M, Amemiya T, Tomii K, Nagano N, Sakaya N, Misoo K, Sato M, Kidera A, Hiroaki H, Shirai T, Kinoshita K, Noguchi T, Ota M. Nucleic Acids Research, 2011, 39:D487-D493.
SAHG, a comprehensive database of predicted structures of all human proteins, and structure prediction pipeline for eukaryotic proteins, Chie Motono, Ryotaro Koike, Kana Shimizu, Matsuyuki Shirota, Takayuki Amemiya, Kentaro Tomii, Nozomi Nagano, Naofumi Sakaya, Kiyotaka Misoo, Miwa Sato, Akinori Kidera, Hidekazu Hiroaki, Tsuyoshi Shirai, Kengo Kinoshita, Tamotsu Noguchi and Motonori Ota., CBI/JSBi2011合同大会「計算科学の拓く新しい生命像」 ポスター JSBi-48 (2011).
Computational overview of GPCR gene universe to support reverse chemical genomics study. Suwa M.,Ono Y. Methods Mol Biol. 2009, 577:41-54. (Acknowledgements:Tatsuya Nishizawa)
タンパク質立体構造予測パイプラインFORTE-SUITE向け網羅的モデリング・構造評価システム(生命情報科学(構造ゲノミクス),ポスター発表,第45回日本生物物理学会年会), 本野 千恵 , 広川 貴次 , 佐藤 美和 , 藤原 康広 , 三十尾 潔高 , 酒谷 尚史 , 富井 健太郎 , 秋山 泰, 生物物理 47(SUPPLEMENT_1), S83, 2007-11-20
立体構造予測実験CASP7に対するとり組み, 富井健太郎,本野千恵,佐藤美和,太田元規,酒谷尚史,藤原康広,三十尾潔高,広川貴次,Paul Horton,秋山 泰: CBRC 2006 予稿集 B5, 28 Sep. 2006.
タンパク質立体構造予測システムFORTE-SUITEの自動化・並列化, 本野千恵,藤原康広,三十尾潔高,佐藤美和,酒谷尚史,富井健太郎,広川貴次,秋山 泰: CBRC 2006 予稿集 G2, 28 Sep. 2006.
eF-site and PDBjViewer: database and viewer for protein functional sites, Kengo Kinoshita, Haruki Nakamura, (Acknowledgements: Dr Hiroyuki Sato), Bioinformatics 20 (8): 1329-1330, 2004
バイオインフォマティクス全般
循環発展的なプロジェクト構造を生むバイオインフォマティクス戦略, 諏訪牧子、小野幸輝, Synthesiology, 2009 Nov, Vol.2 No.4 pp.299-309 (謝辞:西澤達也)
配列解析
Automated classification of alternative splicing and transcriptional initiation and construction of visual database of classified patterns. Nagasaki H., Arita M., Nishizawa T., Suwa M. , Gotoh O. Bioinformatics. 2006 May 15;22(10):1211-6. Epub 2006 Feb 24. PMID: 16500940
6真核生物種における選択的スプライシングと選択的転写開始の種特異性, 長崎 英樹, 有田 正規, 西澤 達也, 諏訪 牧子, 後藤 修, 日本分子生物学会年会講演要旨集 巻:28th ページ:42, 2005年11月25日
Species-specific variation of alternative splicing and transcriptional initiation in six eukaryotes. Nagasaki H., Arita M., Nishizawa T. , Suwa M. , Gotoh O. Gene. 2005 Dec 30;364:53-62. Epub 2005 Oct 10. PMID: 16219431
ヒト遺伝子オルタナティブスプライシングパターンデータベースASTRAの開発, 長崎 英樹, 有田 正規, 西澤 達也, 諏訪 牧子, 後藤 修, 第27回日本分子生物学会年会, 神戸, 2004年12月9日
プロモータ配列の構造解析システム, 矢田 哲士, 十時 泰, 西澤 達也, 中井 謙太, 生物物理 38(Supple 2), S122, 1998-09-07
R
R graphical manualsの開発と今後の展開, 小笠原理, 服部恵美, 三十尾潔高, 統計数理研究所共同研究「Rの整備と利用」研究会, 9 Dec. 2006.
核燃料セラミックス
Study of Fission and High-burnup Induced Restructuring of Nuclear Fuel Ceramics – Applying Computer Science to Investigate Kinetic Process、 Motoyasu Kinoshita, Ying Chen, Yasunori Kaneta, Hua Yun Geng, Misako Iwasawa, Toshiharu Ohnuma, Takashi Ichinomiya, Yasumasa Nishiura, Mitsuhiro Itakura, Jiniichi Nakamura, Kiyotaka Misoo, Soichiro Suzuki and Hans J Matzke、MRS Proceedings、Volume 1043、2007
新クロスオーバ研究「照射・高線量領域の材料挙動制御のための新しいエンジニアリング」分子動力学とモンテカルロ法による計算システムの設計製作、 木下幹康, CHEN Ying, 金田保則, GENG Hua‐Yun, 一宮尚志, 西浦廉政, 三十尾潔高, 鈴木惣一朗, 板倉充洋, 中村仁一, 日本原子力学会春の年会要旨集(CD-ROM) 巻:2007 ページ:N21
材料データベース
材料データベースの統合検索システムの開発、 吉田 健司、真下 忠彰、 情報処理学会誌 2005, 15(3), pp.31-35
プラズマ粒子 (MPI/PVM)
宇宙プラズマ粒子シミュレータKEMPO1の大規模並列化, 秋山 泰,三十尾潔高,大村善治,松本 紘,野口 保: 情報処理学会シンポジウム論文集,巻:98 号:7 ページ:161, 1998.6
Parallelization of Space Plasma Particle Simulation, Yutaka Akiyama, Kiyotaka Misoo, Yoshiharu Omura, Hiroshi Matsumoto, Minoru Saito, Tamotsu Noguchi, Kentaro Onizuka, and Makoto Ando : Lecture Notes on Computer Science, Springer-Verlag, Vol.1336, pp.281-292, 1997.
宇宙プラズマ粒子シミュレーションの並列化, 秋山 泰,三十尾潔高,大村善治,松本 紘,斎藤 稔,野口 保,鬼塚健太郎: 情報処理学会研究報告,97-HPC-66-1, pp.1-6,(つくば), 9 May, 1997.
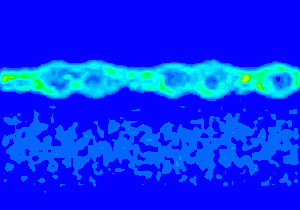 当時作成した動画(3.27MB 位相空間中のプラズマ粒子分布の時間変化、並列化したKEMPOコードによる高速計算)
当時作成した動画(3.27MB 位相空間中のプラズマ粒子分布の時間変化、並列化したKEMPOコードによる高速計算)
