IPABコンテスト「コンピュータで薬のタネを創る」に参加し、グランプリNEC賞を受賞しました。コンテストのテーマは、チロシンキナーゼの一種であるヒトc-Yesに対する阻害剤の探索です。この記事では、医薬品候補化合物の探索に用いた手法と技術をご紹介します。
手法の概略
今回、候補化合物の探索のために採用したのは、阻害剤となり得る化合物の性質に着目したligand-based virtual screeningに分類される方法です。c-Yesに対する阻害活性が実験的に測定されている化合物の情報をもとに、220万種類の化合物から阻害剤として有望な化合物の絞り込みを行いました。具体的な手順を以下に示します。
- c-Yesに対する阻害活性が測定されている839化合物を学習用データセットとして、機械学習により回帰モデルを構築しました。
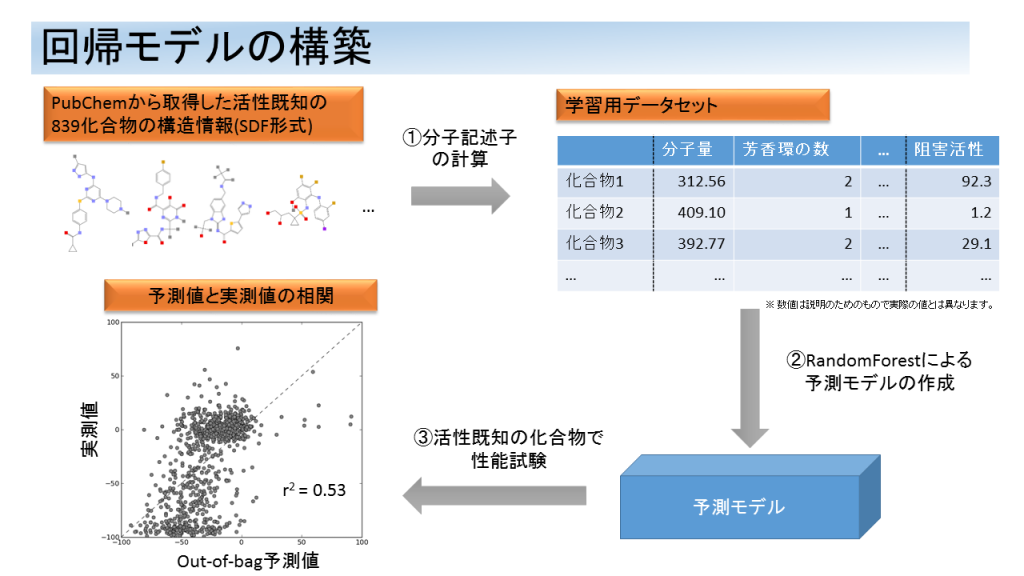
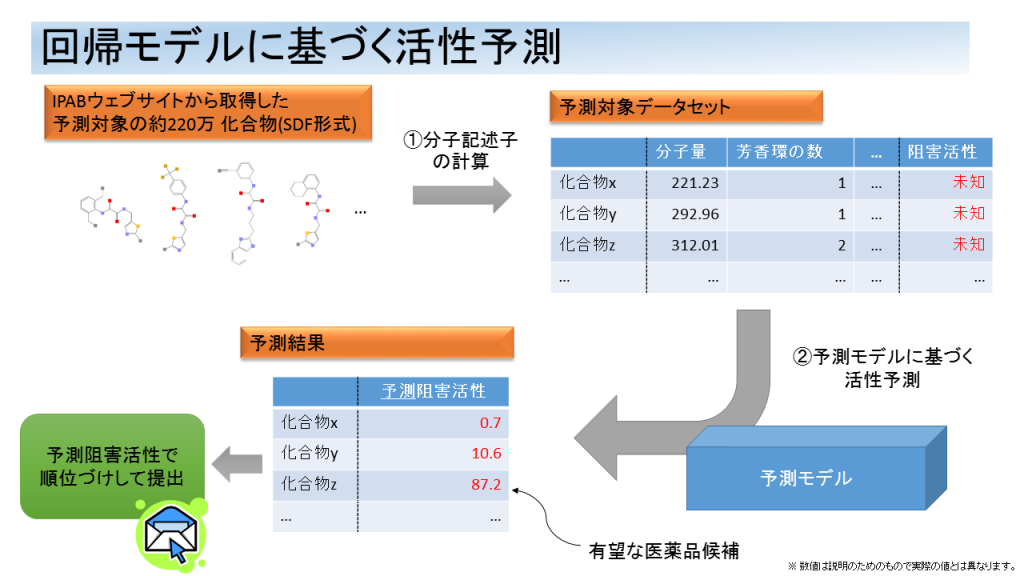
2. 構築された回帰モデルに基づいて、化合物ライブラリに含まれる約220万化合物について、阻害活性の予測を行いました。
この結果、高い阻害活性が予測された上位120化合物を阻害剤候補として提案しました。
オープンソース・ライブラリを利用したPythonによる科学技術計算
候補化合物の探索に使用したソフトウェアは、すべてオープンソースソフトウェアです。特に中核部分は、いくつかのオープンソースのライブラリを利用し、Python言語で開発した独自のプログラムを使用しました。Pythonは科学技術計算のためのライブラリが充実しているため、ケムインフォマティクスから機械学習までを簡単なコードで完結できます。本コンテストで使用したライブラリは以下の通りです。
| NumPy | 数値計算のためのライブラリ。ベクトルや行列の演算がPythonだけで書くより高速に行えます。 |
| Pandas | データ解析のためのライブラリ。データフレームを用いたR言語ライクなデータの取り扱いが可能になります。 |
| matplotlib | グラフ描画のためのライブラリ。散布図やヒストグラムを初めとする様々な形式のグラフに対応しカスタマイズも可能です。 |
| scikit-learn | 機械学習のためライブラリ。今回使用したRandomForest以外にもサポートベクターマシン、K平均法等のアルゴリズムをサポートしています。 |
| RDKit | ケモインフォマティクスのためのライブラリ。SDF形式の化学構造情報の処理や分子記述子の計算に対応。C++向けAPIも用意されています。 |

